Doktora Adayı: Müslüm Kaan Arıcı
EABD: Tıp Bilişimi
Tarih: 22.01.2024 / 14:00
Yer: A-108
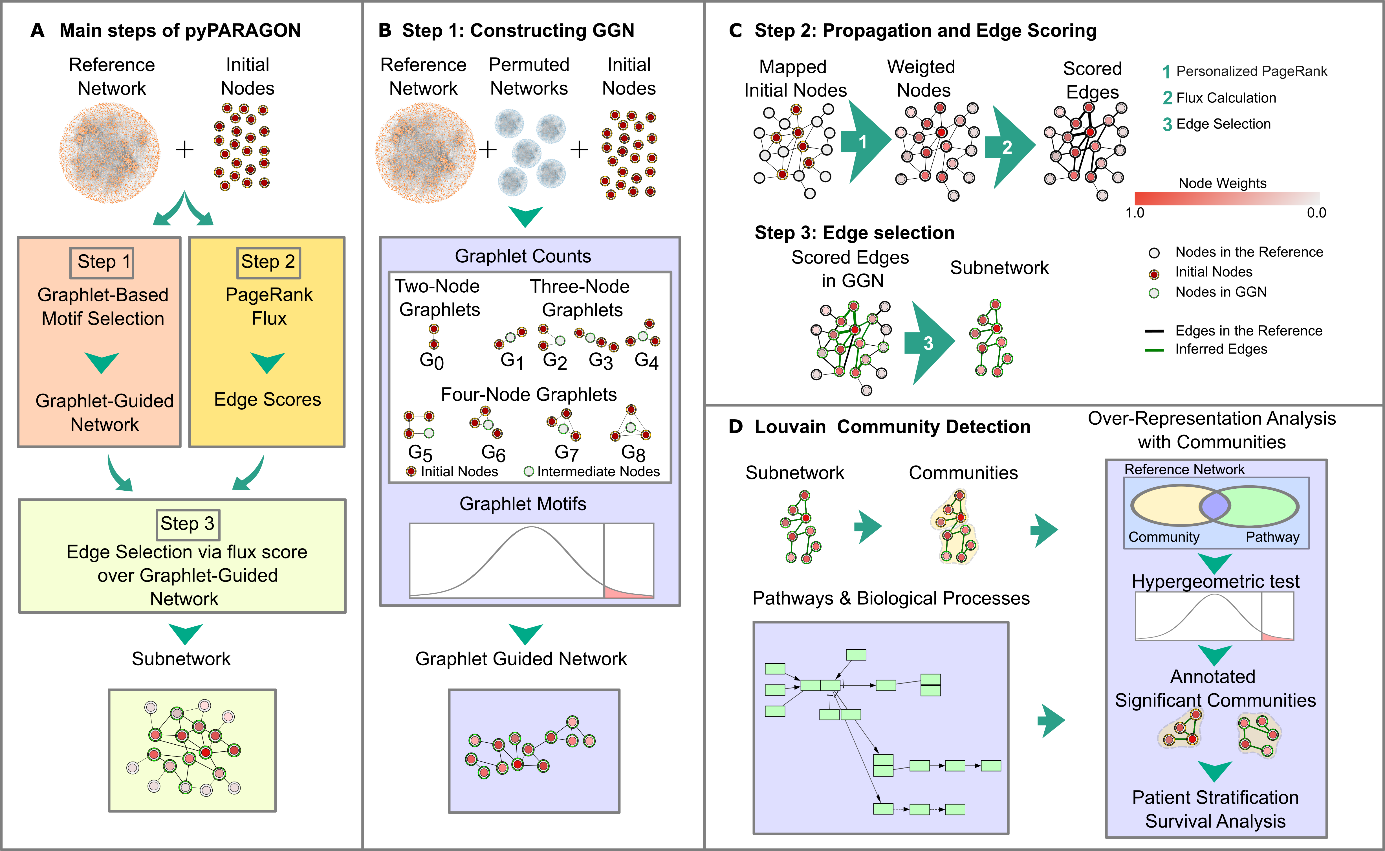
Özet: Omik teknolojileri, hastalık durumları, hastalar ve ilaç bozuklukları gibi çeşitli biyolojik konularda moleküler bilgi sağlar. Ağ varsayım ve yeniden yapılandırma yöntemleri, belirli biyomoleküler etkileşimler ve faaliyetlerle bağlamlı ağlar oluşturmak için birkaç omics veri kümesi kullanırlar. Bu tezde referans ağındaki eksiklikleri tespit etmek için yulaklar, üç boyutlu yapılar ve academic yayın sayımları gibi çeşitli bilgi türlerinde referans ağlarının kapsamlarını karşılaştırarak bir değerlendirme analizi yaptık. Buna ek olarak, yeniden yapılandırma algoritmalarının eksikliklerini sinyal yollarını sonlandırarak inceledik. Kontekstüelleştirilmiş ağ varsayımında bazı iddialı sorunlar tespit ettik: (i) Omics veritabanlarının hitleri referans ağlarında azdır (ii) Yorumlama yöntemleri, multi-omics veritabanlarını değerlendirirken omics veri kümelerinde önemli hitler bağlayan gizli bilgiyi kaçırabilir. iii) Referans ağındaki fazla çalışılmış proteinler bağlamda önyargı ile birlikte gelir. (iv) Yüksek derecede bağlantılı düğümler (hublar) ağlarda belirli olmayan ve gürültülü etkileşimlere yol açarlar. Bu sorunları çözmek için, multi-Omics veri entegrasyonu için pyPARAGON (PAgeRAnk-flux on Graphlet-guided network for multi- Omics data integratioN) geliştirdik. Performans testlerinde, pyPARAGON ağ motiflerini kullanarak, kanser sinyal yollarının yeniden yapılandırılması ve bağlamsal kanser modellerinin bulunmasıyla sinyal ağlarında hassasiyetini artırarak ve alakasız etkileşimleri azalmıştır. Ayrıca, örnek çalışmlarda, pyPARAGON, tümöre özgü ağlarda önemli biyolojik sürecleri ve yulakları cıkartarak ve kanser ve nörolojik gelişim bozuklukları modellerinde ortak yulakları ve farklı sinyal güçlerini bularak umut verici bir performans göstermiştir.