Yüksek Lisans Adayı: Elif Bozlak
EABD: Biyoenformatik
Tarih: 29.07.2019 / 11:00
Yer: A-212
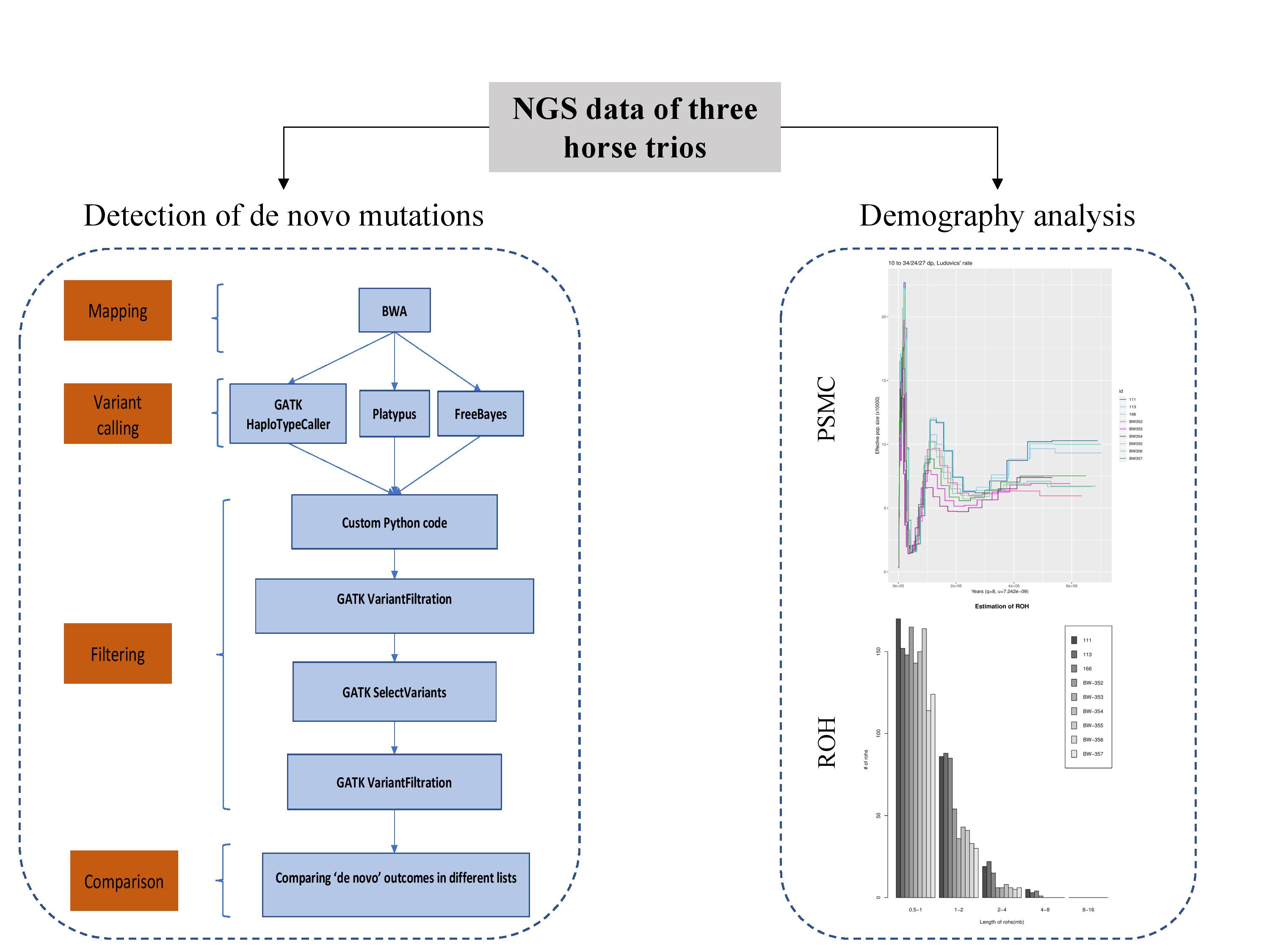
Özet: De novo mutasyonlar ebeveynlerde görülmezken yavruda ortaya çıkan ve Mendel kalıtım kurallarına uymayan mutasyonlardır. Popülasyonların evrimsel tarihlerinin anlaşılmasında yardımcı oldukları için de novo mutasyonaların sayılarının tespit edilmesi genetik çalışmalar için önemlidir. Bu tezde evcil atlarda bir jenerasyonda ortaya çıkan de novo mutasyonları tespit etmeyi ve atların demografik tarihleri üzerine tahminler yapmayı hedefledik. Çalışmada üç farklı at türü (Lipizzaner, Noriker ve Haflinger) için yeni nesil sekanslama teknolojisi ile üretilen üçleme DNA sekans verilerini kullandık. Ham verinin kalite kontrolü ve hizalanmasından sonra, üç farklı varyant çağırma algoritması kullanarak genomik varyantları çağırdık. Tüm varyantları kalitelerine göre filtreledik ve seçilen 50 varyantı Sanger sekanslama ile laboratuarda test ettik. Test edilen varyantların yaklaşık olarak %40’ı valide edildi. Yüksek okuma derinliğine sahip Lipizzaner (n=13) türündeki gerçek pozitif sayısını yüksek, düşük okuma derinliğine sahip Noriker (n=3) ve Haflinger (n=5) türlerindekileri ise daha düşük sayıda bulduk. Sonuçlar gerçek pozitif de novo mutasyonların tespit edilmesinde okuma derinliğinin önemini gösterdi. Ek olarak elimizdeki at popülasyonlarının demografik tarihleri hakkında tahmin yürütmek için PSMC modeli oluşturduk ve ROH analizi yaptık. PSMC ve ROH sonuçları önceki çalışmalarla uyumlu sonuçlar verdi. Sonuç olarak tüm genom sekanslama verisi ile de novo mutasyon tespiti ve popülasyon demografisi tahmini yapabilmek için gereken minimum veri okuması ve kalitesi hakkında fikir sahibi olduk.